李超教授团队在Scientific Data上发布泰山螭霖鱼染色体水平基因组
2025-08-16 13:16:39 作者:李超 来源:宣传部 浏览数:0
近日,青岛农业大学李超团队在Scientific Data(IF=6.932)上发表了题为“Achieving Chromosome-Level Genome Assembly of Onychostoma macrolepis with PacBio Sequencing and Hi-C Technologie”的研究论文,在染色体水平上解析了泰山螭霖鱼的基因组,为今后的泰山螭霖鱼研究提供了宝贵的资源。

泰山螭霖鱼学名多鳞白甲鱼(Onychostoma macrolepis)是一种新兴的经济鱼类,主要分布于我国的长江、淮河、渭河和海河水系。因其肉质细嫩、味道鲜美且具有药用价值,其价格是普通鲤鱼(Cyprinus carpio)的近20倍。近几十年来,由于过度捕捞和环境污染,野生种群数量急剧下降,目前已被列入《国家重点保护野生动物名录》,成为国家二级保护动物。关于泰山螭霖鱼的研究受到了越来越多的关注。
研究人员利用PacBio三代测序、Hi-C技术和二代测序策略,并使用ONT超长读长进行间隙填充,在染色体水平上组装了泰山螭霖鱼的高质量参考基因组。组装的基因组大小为944.02 Mb,contig N50长度为32.36 Mb,大约99.15% 的组装序列锚定在25条染色体上,BUSCO和CEGMA的评估结果分别为97.9%和95.56%,QV值为48.86,Illumina双端测序读段的重比对覆盖率为99.59%,说明该基因组的组装质量较高。
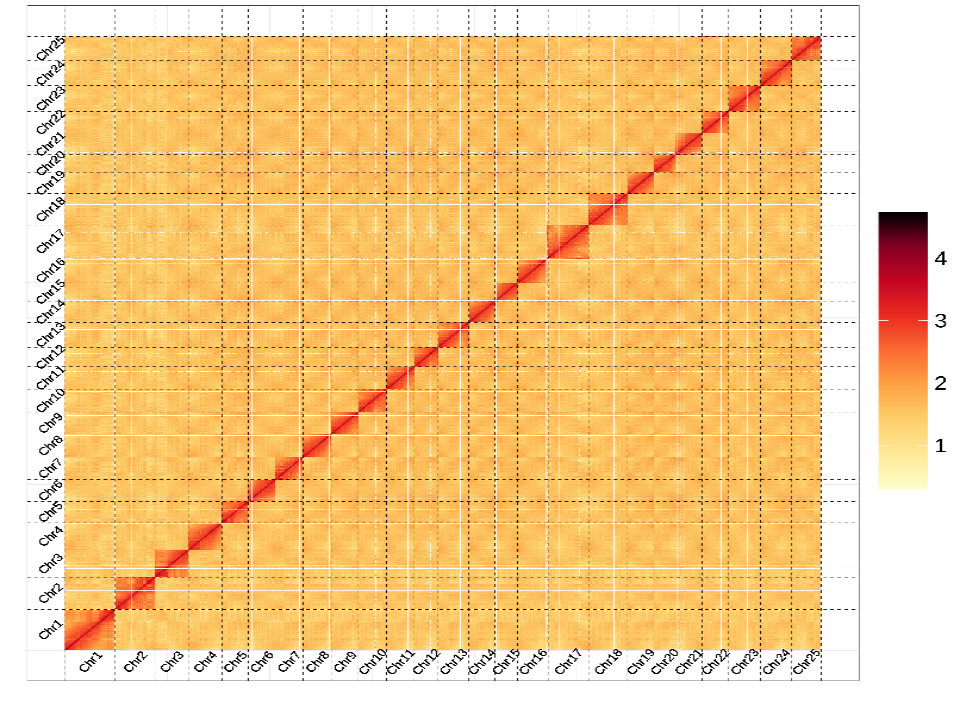
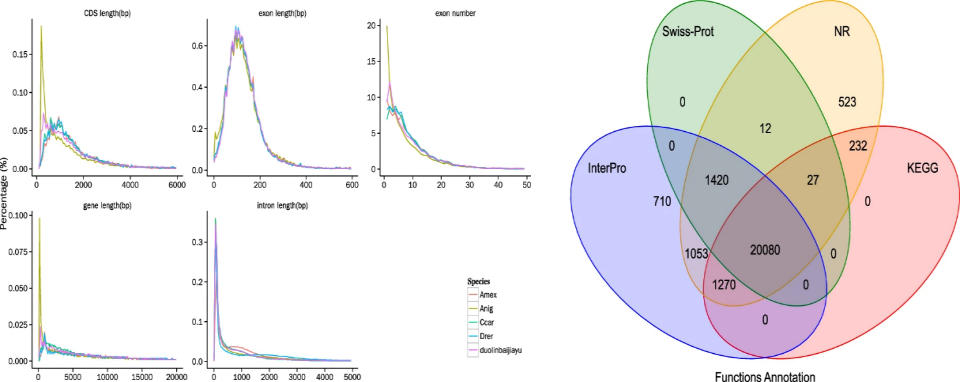
通过整合同源预测、从头预测和RNA-Seq辅助预测三种策略,结合多工具流程和跨数据库功能注释,最终在泰山螭霖鱼基因组鉴定出了26,294 个蛋白质编码基因,其中96.30% 的基因进行了功能注释。
该研究由青岛农业大学海洋科学与工程学院联合泰安市农业科学院完成。青岛农业大学海洋科学与工程学院李超教授团队博士生张璐为该论文第一作者。李超教授为通讯作者,朱颖老师和曹敏副教授参与了该研究工作。该工作得到了山东省鱼类产业技术体系(SDAIT-12-02)和山东省自然科学基金(ZR2024QC027)的支持。
- 上一篇: 没有了
- 下一篇: 海洋学院章晋勇教授课题组在利用木质纤维素底物高效转化微生物油脂和类胡萝卜素方面取得新进展
邮编:266237 电话:0532-86550511 邮箱:hyxy@qau.edu.cn
地址:即墨区鳌山卫街道问海路17号青岛农业大学海洋科学与工程学院
鲁ICP备13028537号 -5 鲁公网安备 37021402000104号青岛市互联网违法信息举报中心 网络信息员:0532-86550515





